FInE
Fonctionnalité de l'écosystème intestinal
Le microbiote intestinal humain, tel que nous le connaissons aujourd’hui, semble être très complexe. Afin de progresser vers une compréhension holistique du microbiote intestinal, y compris de sa composante non cultivable, encore majoritaire à ce jour, nous avons contribué au développement d’une nouvelle et puissante approche, la métagénomique, qui a permis des progrès rapides dans la caractérisation de la diversité génomique et génétique du microbiote intestinal. Par exemple, le génome microbien (microbiome) de chaque individu contient ~28 fois plus de gènes que le génome humain. Le microbiote est d’ailleurs considéré comme un organe hôte à part entière.
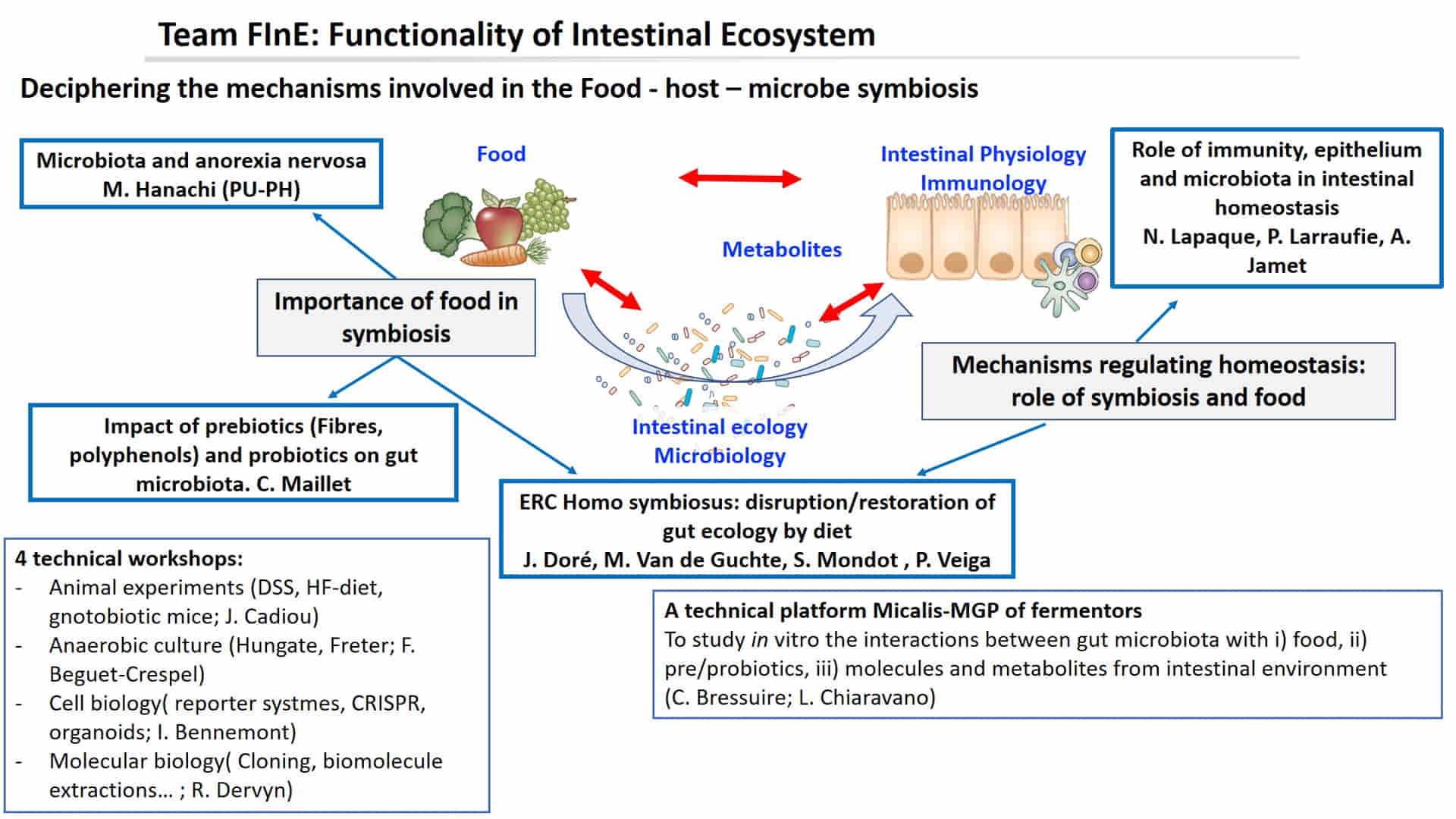
L’équipe FInE vise à approfondir la compréhension actuelle du rôle fondamental joué par le microbiote dans la santé humaine. Notre objectif est de mieux comprendre les mécanismes de la symbiose homme-microbe et l’influence de notre alimentation, en particulier l’importance des fibres, des polyphénols et des probiotiques, afin de définir des stratégies pour sa modulation. FInE est une équipe mixte rattachée aux départements MICA et AlimH de l’INRAE. Elle est étroitement liée à MetaGenoPolis puisqu’elle a contribué à l’émergence de deux de ses plateformes (SAMBO et MétaFun).
Axes de recherche
Anorexie mentale et microbiote
Impact de l'alimentation, des probiotiques... sur l'écologie intestinale
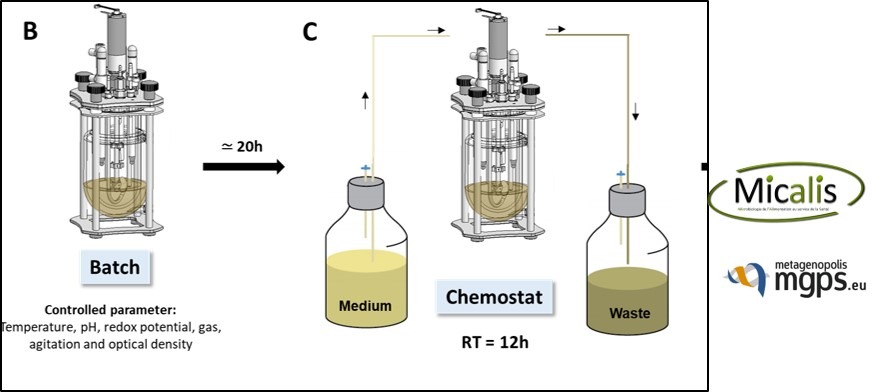
- Richesse de la diversité (ARNr 16S), métabolites (SCFA…), métagénomique quantitative, test sur système rapporteur de cellules épithéliales et tests enzymatiques, évolution des bactéries (pathogènes ou commensales).
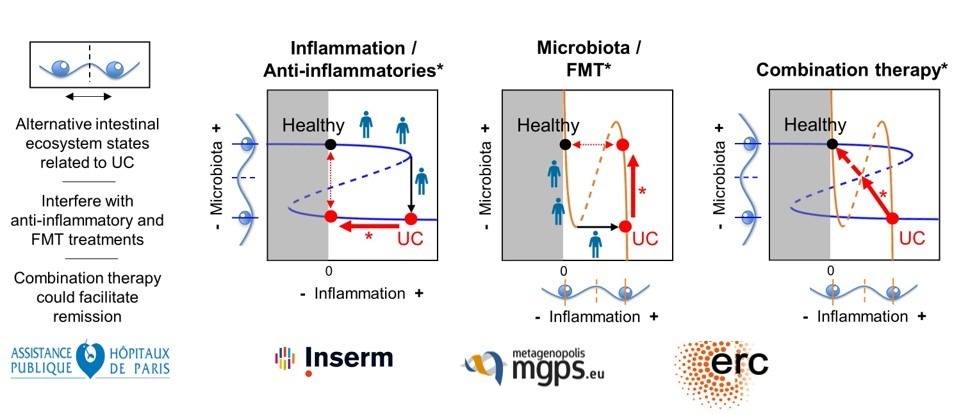
Un nouveau concept de transition critique dans l'écosystème intestinal.
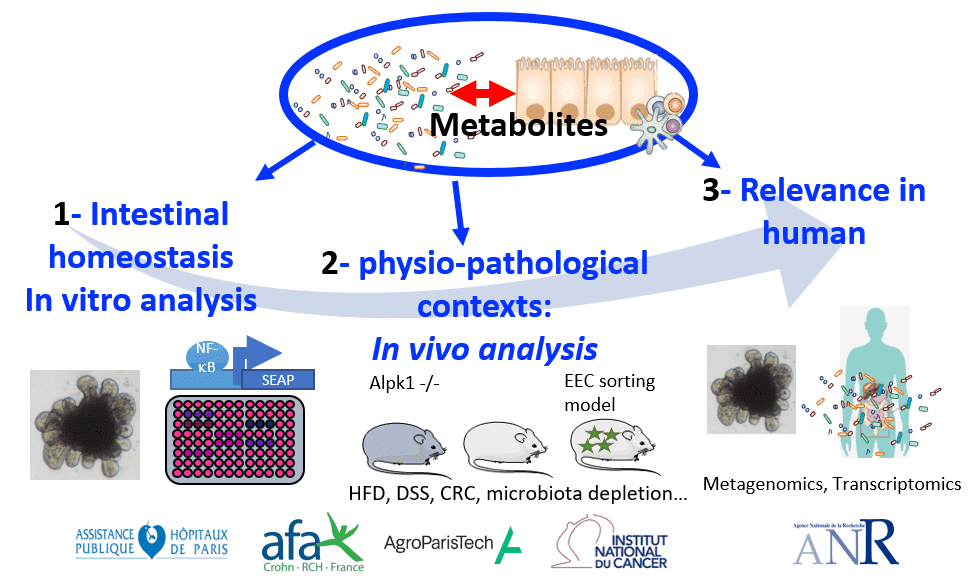
Réponse de l'hôte aux modulations microbiennes
- Role of innate immune receptors on intestinal homeostasis (obesity, colitis and colorectal cancer).
- Role of the microbiota on enteroendocrine cells plasticity, impact on gut hormones
- Tools used: cell biology (reporter cell lines, CRISPR-Cas9 gene editing, organoids), in vivo analysis (KO and mouse models for cell sorting), transcriptomics, peptidomics
Membres de l'équipe
Alumni
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
Faits marquants
- Martin-Gallausiaux C, Salesse L, Garcia-Weber D, Marinelli L, Beguet-Crespel F, Brochard V, Le Gléau C, Jamet A, Doré J, Blottière HM, Arrieumerlou C, Lapaque N (2024) Fusobacterium nucleatum promotes inflammatory and anti-apoptotic responses in colorectal cancer cells via ADP-heptose release and ALPK1/TIFA axis activation. Gut Microbes 16(1).
- Ngom SI, Maski S, Rached B, Chouati T, Oliveira Correia L, Juste C, Meylheuc T, Henrissat B, El Fahime E, Amar M, Béra-Maillet C (2023) Exploring the hemicellulolytic properties and safety of Bacillus paralicheniformis as stepping stone in the use of new fibrolytic beneficial microbes. Scientific Reports 2023 13:1 13(1):1–16.
- Martin-Gallausiaux C, Garcia-Weber D, Lashermes A, Larraufie P, Marinelli L, Teixeira V, Rolland A, Béguet-Crespel F, Brochard V, Quatremare T, Jamet A, Doré J, Gray-Owen SD, Blottière HM, Arrieumerlou C et al (2022) Akkermansia muciniphila upregulates genes involved in maintaining the intestinal barrier function via ADP-heptose-dependent activation of the ALPK1/TIFA pathway. Gut Microbes 14(1).
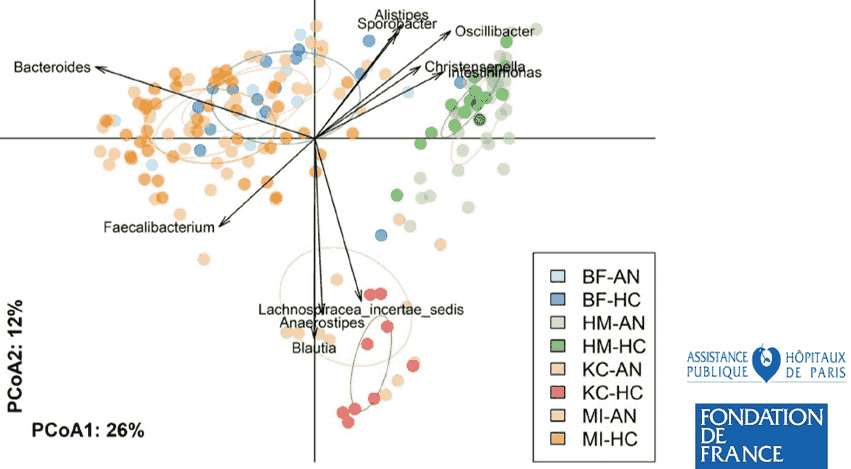
- Mondot S, Lachkar L, Doré J, Blottière HM, Hanachi M (2022) Roseburia, a decreased bacterial taxon in the gut microbiota of patients suffering from anorexia nervosa. European journal of clinical nutrition 76(10):1486–1489.
- Di Lodovico L, Mondot S, Doré J, Mack I, Hanachi M, Gorwood P (2021) Anorexia nervosa and gut microbiota: A systematic review and quantitative synthesis of pooled microbiological data. Progress in Neuro-Psychopharmacology and Biological Psychiatry 106
- van de Guchte M, Mondot S, Doré J (2021) Dynamic Properties of the Intestinal Ecosystem Call for Combination Therapies, Targeting Inflammation and Microbiota, in Ulcerative Colitis. Gastroenterology 161(6):1969-1981.e12.
- Martin-Gallausiaux C, Marinelli L, Blottière HM, Larraufie P, Lapaque N (2021) SCFA: mechanisms and functional importance in the gut. Proceedings of the Nutrition Society 80(1):37–49.
- Van de Guchte M, Burz SD, Cadiou J, Wu J, Mondot S, Blottière HM, Doré J (2020) Alternative stable states in the intestinal ecosystem: proof of concept in a rat model and a perspective of therapeutic implications. Microbiome 8(1):153.
Voir toute la collection HAL ici
Financements externes
- ANR EnteroFructose (2024-2027) : Coordinatrice Véronique Douard
- ANR Gut-Alpk1 (2022-2025) : Nicolas Lapaque
- ANR IMAT (2021-2024) : Coordinateur Maude LeGall (INSERM)
- PLBIO-INCA (2021-2024) : Nicolas Lapaque
- ERC Advanced Homo-Symbosius (2019-2023) : Joël Doré
- EU FP7 Microb-Predict (2019-2026) Coordinateur : Jonel Trebicka (EF CLIF, Barcelone)
- ANR LUMI (2019-2022) Coordinatrice : Sylvia Cohen-Kaminsky (INSERM)
Plusieurs projets collaboratifs avec des industriels sont également en cours DuPont, Sanofi et Roquette.
GRANDS PROJETS passés auxquels nous avons contribué :
- ANR LUMI (2019-2022) Coordinatrice : Sylvia Cohen-Kaminsky (INSERM)
- ANR FunAMetaGen (2015-2019) Coordinateur : Laurent Chene (Entérome)
- ANR PigletBiota (2014-2018) Coodinateur : J Estellé (INRA – Gabi)
- ANR ProteoCardis (2016-2020) Coordinatrice : Catherine Juste
- EU FP7 MetaCardis (2012-2018) Coordinatrice : K Clément (ICAN)
- EU FP7 IHMS (2011-2015) Coordinateur : SD Ehrlich (MGP)
- EU FP7 METAHIT (2008-2012) Coordinateur : SD Ehrlich (MGP)
- EU FP7 Cross-Talk (2008-2012) Coordinateurs : E Maguin (Ife – Micalis) & HM Blottière (FInE – Micalis)
- ANR SUSFLORA (2011-2014) Coordinatrice : C Rogel-Gaillard (INRA – Gabi)
- ANR Surfing (2011-2014) Coordinateurs : G Jan (STLO – INRA) & M van de Guchte (Ife – Micalis)
- ANR SFBIMPRO (2010-2013) Coordinatrice : V Gaboriau-Routhiau (INSERM – Necker)
- ANR EPIFLORE (2013-2016) Coordinatrice : MJ Butel (Université Paris Descartes)
- ANR FunMetaGen (2012-2014) Coordinateur : HM Blottière (FInE – Micalis)
- ANR MicroObes (2008-2012) Coordinateur : J Doré (FInE – Micalis)
- Risk OGM CRYMUC (2012-2016) Coordinatrice : C. Nielsen-Leroux (GME – Micalis)
- INRA MEM MetaScreen (2011-2013) Coordinatrice : G Veronese (LISBP – INRA
- INRA MEM OBOMICS (2012-2014) Coordinatrice : C Juste (FInE – Micalis)