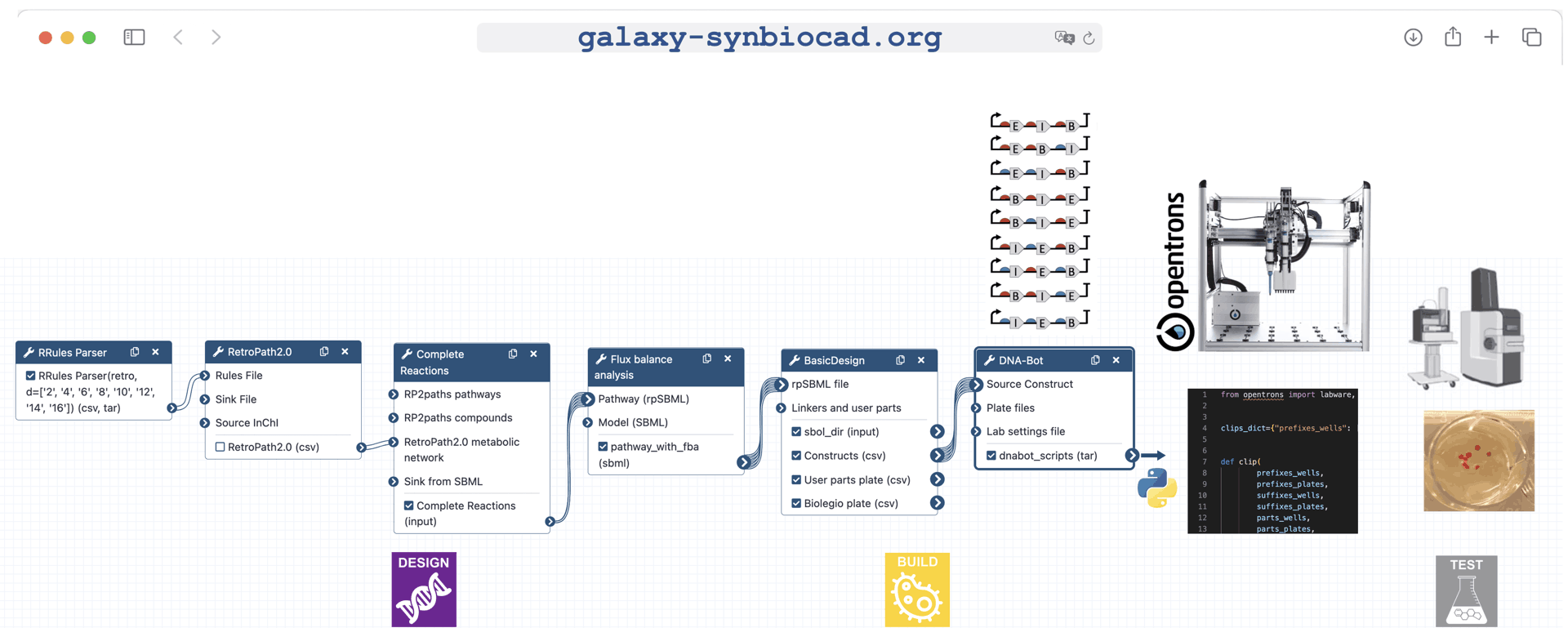
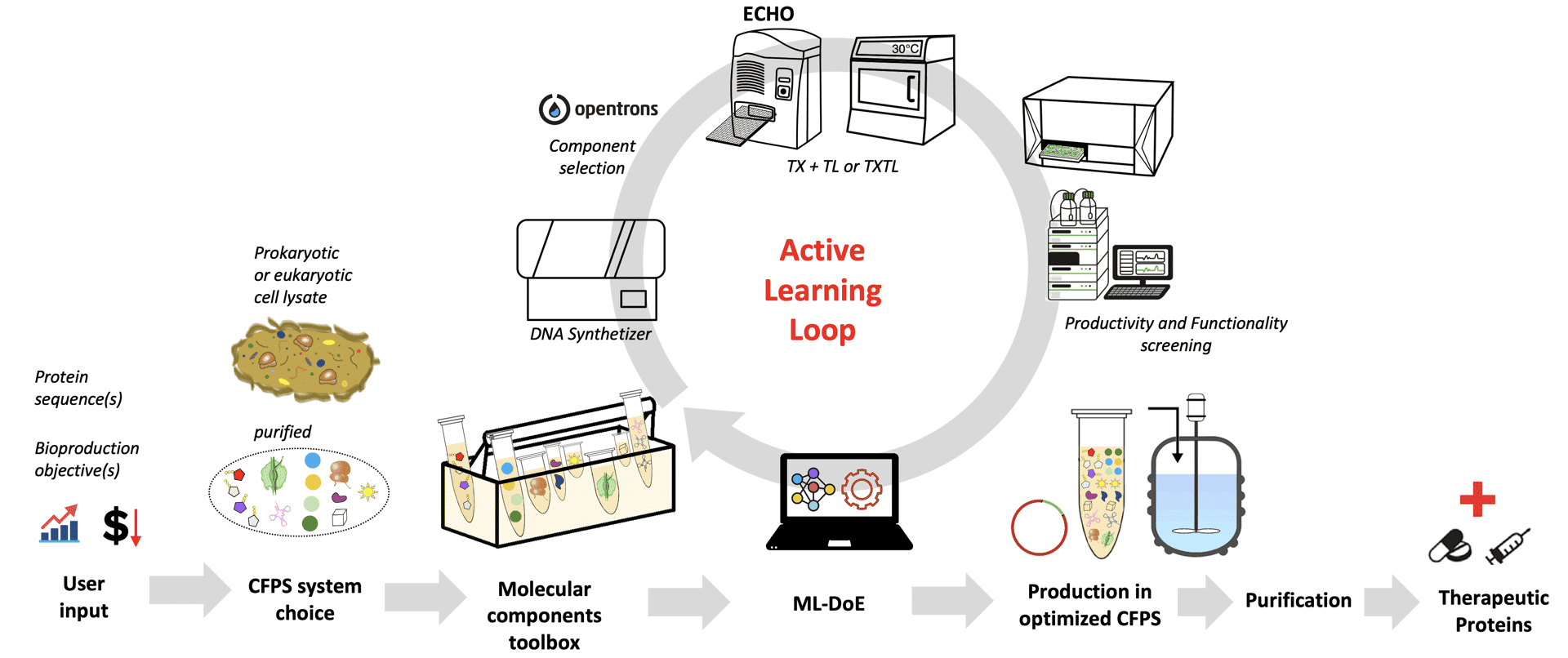
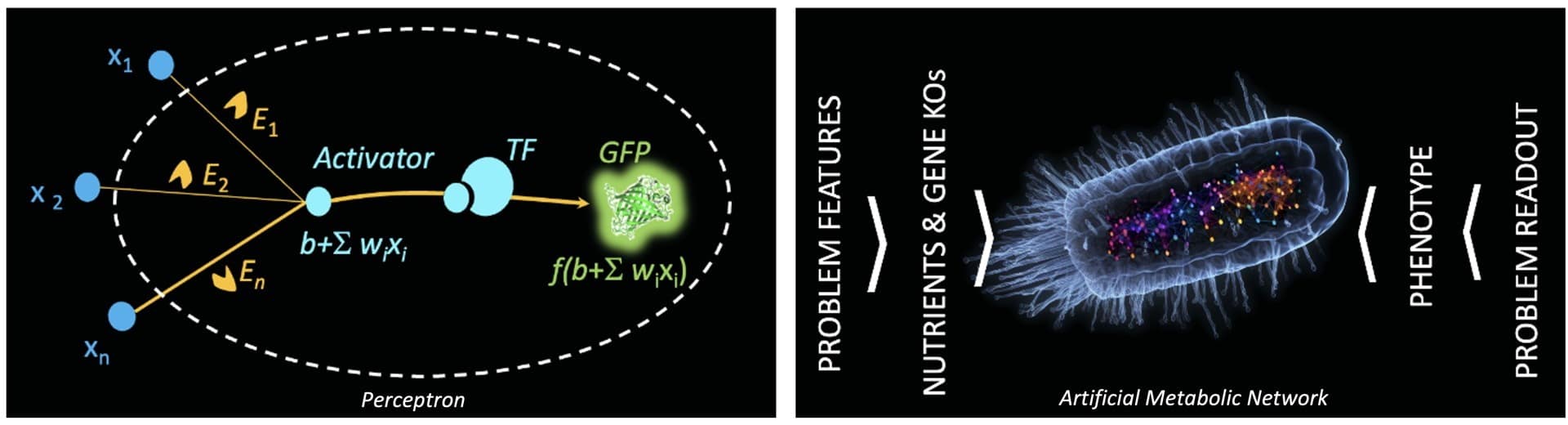
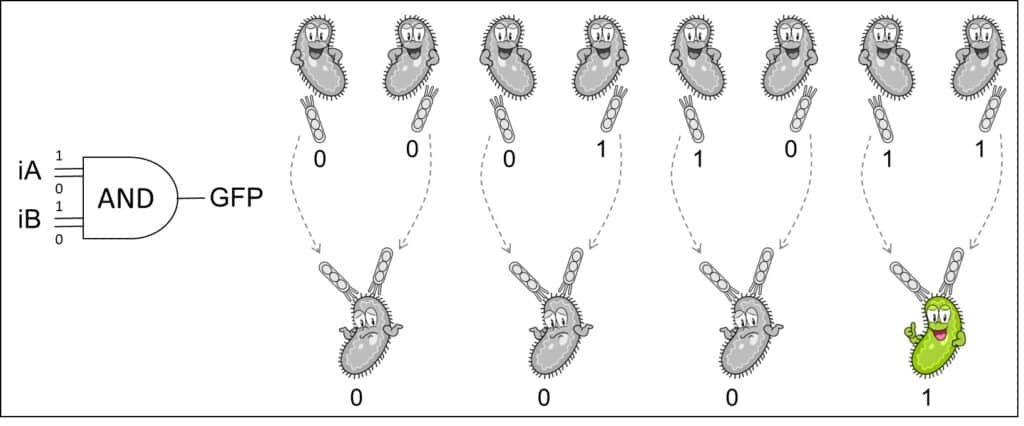
-
- Combac • Communication Bacterienne
- DynPhages • Dynamique du génome des bactériophages
- B3D • Biofilms et communautés spatialement organisées
- CPE • Commensalisme et pathogenèse des entérocoques
- MicrobAdapt • Déterminants de l’adaptation microbienne
- Paroi • Dynamique de la paroi cellulaire bactérienne
- EpiMiC • Epigénétique et microbiologie cellulaire
- GME • Génétique Microbienne et Environnement
- BaPS • Bactéries pathogènes et santé
- PIMs • Pathogènes, immunité et microbiote
- Combac • Communication Bacterienne
- DynPhages • Dynamique du génome des bactériophages
- B3D • Biofilms et communautés spatialement organisées
- CPE • Commensalisme et pathogenèse des entérocoques
- MicrobAdapt • Déterminants de l’adaptation microbienne
- Paroi • Dynamique de la paroi cellulaire bactérienne
- EpiMiC • Epigénétique et microbiologie cellulaire
- GME • Génétique Microbienne et Environnement
- BaPS • Bactéries pathogènes et santé
- PIMs • Pathogènes, immunité et microbiote
- ChemSyBio • Biochimie et Biologie Synthétique
- ProbiHôte • Interactions des Bactéries Commensales et Probiotiques avec l’Hôte
- NutriPhage • Dynamique du virome intestinal en fonction du régime alimentaire
- AMIPEM • Alimentation, microbiote intestinal, maladies cérébrales et métaboliques
- FME • Écologie microbienne alimentaire
- FInE • Fonctionnalité de l’écosystème intestinal
- MIHA • Interactions du microbiote avec l’homme et l’animal
- PhylHom • Phylogénie et physiologie du microbiome humain
- ChemSyBio • Biochimie et Biologie Synthétique
- ProbiHôte • Interactions des Bactéries Commensales et Probiotiques avec l’Hôte
- NutriPhage • Dynamique du virome intestinal en fonction du régime alimentaire
- AMIPEM • Alimentation, microbiote intestinal, maladies cérébrales et métaboliques
- FME • Écologie microbienne alimentaire
- FInE • Fonctionnalité de l’écosystème intestinal
- MIHA • Interactions du microbiote avec l’homme et l’animal
- PhylHom • Phylogénie et physiologie du microbiome humain
-