FME
Écologie microbienne alimentaire
Comprendre l’écologie microbienne de la chaîne alimentaire : vers une alimentation plus durable et plus saine.
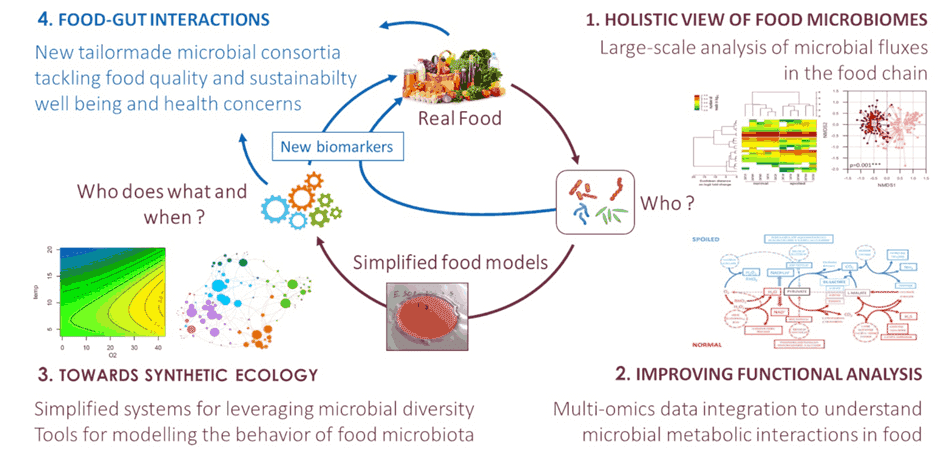
Le laboratoire FME concentre ses recherches sur la compréhension des mécanismes par lesquels les bactéries et les champignons interagissent avec les produits alimentaires au cours de l’altération et de la fermentation. En particulier, notre équipe tente de comprendre comment la diversité microbienne alimentaire peut être exploitée pour concevoir des solutions microbiennes pour une production d’aliments fermentés plus durable et plus saine. Nous développons une analyse fondamentale de l’écologie microbienne alimentaire, y compris la métagénomique, la métatranscriptomique et la métabolomique, la génomique comparative et les analyses globales des fonctions métaboliques réalisées par les différentes espèces microbiennes. Comme le montre la figure, cette stratégie scientifique repose sur quatre axes de recherche. Une stratégie fortement interdisciplinaire combine les résultats et les approches de l’écologie microbienne, de l’analyse des données omiques à haut débit et de la biologie computationnelle. Notre activité scientifique crée une base solide pour développer des programmes innovants en aval avec des acteurs industriels dans le domaine des aliments fermentés. Le laboratoire FME gère également les micro-fermenteurs Ferment-du-Futur BioLector XT dans le cadre de la plateforme PIAM pour l’étude des interactions métaboliques microbiennes dans les modèles alimentaires. Plus d’infos sur notre laboratoire et nos différentes activités ?

Marie Champomier-Vergès & Stéphane Chaillou
Axes de recherche
Vue holistique des microbiomes alimentaires dans la chaîne alimentaire
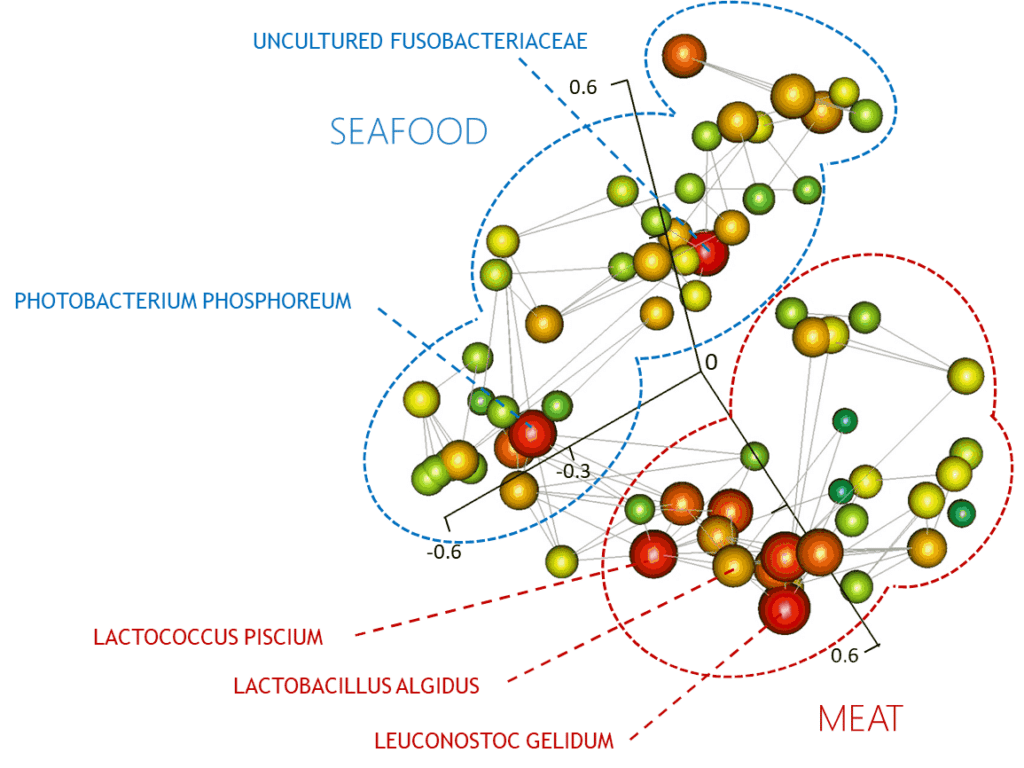
Nos recherches s’appuient sur deux approches. La première consiste en de vastes et ambitieuses enquêtes d’échantillonnage visant à collecter des centaines à des milliers de points de données sur un environnement industriel ou un type d’aliment donné. L’analyse métagénétique (par exemple avec les gènes ARNr 16S, les marqueurs ITS ou gyrB) et l’analyse discriminante multivariée sont ensuite effectuées pour comprendre comment les paramètres de production alimentaire façonnent les communautés microbiennes. Notre travail a démontré le potentiel de ce type de données (obtenues en collaboration avec des partenaires industriels) pour le développement d’outils prédictifs et la caractérisation des étapes de transformation qui peuvent influencer la composition et l’activité microbienne dans le contexte de la production et du stockage des aliments. La seconde approche repose sur une analyse fonctionnelle approfondie des microbiomes alimentaires, combinée à l’utilisation de la métagénomique et de l’analyse génomique et fonctionnelle comparative de nouvelles souches et espèces isolées des aliments. Dans ce contexte, nous mettons en place un outil d’étude métagénomique facile d’accès intégrant des données génomiques publiques (gènes, fonctions…) avec des données métagénomiques (profils de gènes et variations de nucléotides déduits du séquençage shotgun) et des ensembles de données écologiques et technologiques.
Améliorer l'analyse fonctionnelle des interactions métaboliques dans l'alimentation
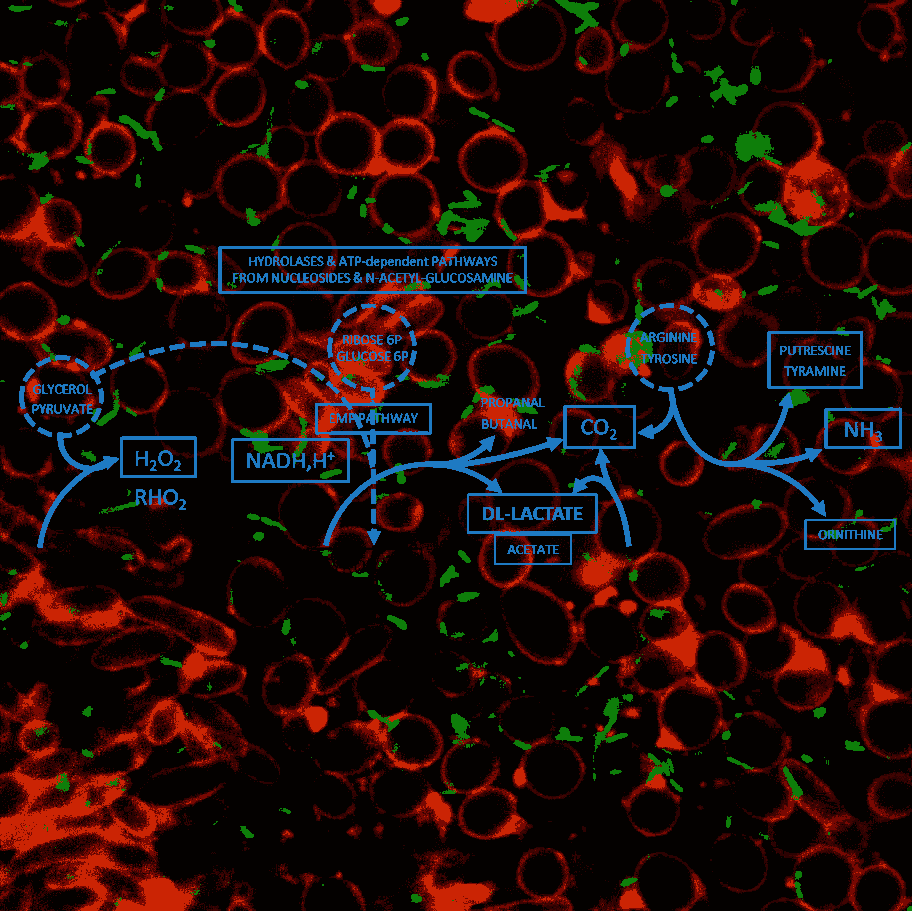
Les interactions métaboliques entre les espèces microbiennes sont caractérisées par des liens entre les espèces dominantes et sous-dominantes ou des populations successives. Les populations peuvent être structurées dans l’espace et peuvent également être influencées par des facteurs environnementaux. La simple cartographie des populations et des métagénomes n’est pas suffisante pour déchiffrer les interactions sous-jacentes qui opèrent au sein des communautés microbiennes pendant leur croissance dans les aliments. Notre approche scientifique se concentre sur le développement d’analyses métatranscriptomiques à l’aide d’outils informatiques et de bases de données spécifiquement développées. La mise en correspondance des transcriptomes avec une base de données contenant plusieurs centaines de fonctions métaboliques fournit une mesure détaillée des fonctions métaboliques exprimées à différents niveaux de la communauté, de l’espèce à l’ensemble du consortium microbien. De cette manière, nous sommes en mesure d’interpréter les interactions qui ont lieu à différentes échelles. Notre travail consiste également à utiliser des données métabolomiques non ciblées et à intégrer ces données multivariées à l’aide d’outils statistiques. Jusqu’à présent, nos travaux se sont concentrés sur l’étude des interactions associées à l’altération des produits carnés et des fruits de mer et sur la manière dont la réduction des conservateurs chimiques (sel, nitrite, lactate) pourrait influencer ces interactions.
Vers une écologie microbienne alimentaire synthétique

Interactions entre les aliments et l'intestin

Les microbes alimentaires peuvent avoir des effets positifs ou négatifs importants sur la production de divers aliments fermentés, ainsi que sur l’intestin humain et la santé digestive.Bien qu’une grande diversité de communautés microbiennes puisse être identifiée dans de nombreux aliments fermentés, on manque d’informations significatives sur leur contribution à la santé humaine. Nous cherchons à comprendre comment la diversité des microbes alimentaires contribue à un microbiome intestinal sain et comment la consommation à long terme d’aliments fermentés a un impact bénéfique sur la santé.
Notre objectif est d’utiliser les données sur les aliments et le microbiome intestinal pour permettre une conception plus pertinente et orientée vers la santé des consortiums microbiens pour les aliments fermentés et d’identifier les biomarqueurs liés à l’effet sur la santé de ces aliments fermentés.
Membres de l'équipe
Alumni
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
- Liste à puce
Faits marquants
- Poirier, S. et al. Holistic integration of omics data reveals the drivers that shape the ecology of microbial meat spoilage scenarios. Frontiers in Microbiology 14, 1286661 (2023).
- Kothe, C. I., Bolotin, A., Kraïem, B.-F., Dridi, B. & Renault, P. Unraveling the world of halophilic and halotolerant bacteria in cheese by combining cultural, genomic and metagenomic approaches. International journal of food microbiology 358, 109312 (2021).
- Zagdoun, M., Coeuret, G., N’Dione, M., Champomier-Vergès, M.-C. & Chaillou, S. Large microbiota survey reveals how the microbial ecology of cooked ham is shaped by different processing steps. Food Microbiology 91, 103547 (2020).
- Verplaetse, E. et al. Heme Uptake in Lactobacillus sakei Evidenced by a New Energy Coupling Factor (ECF)-Like Transport System. Applied and environmental microbiology 86, (2020).
- Terán, L. C. et al. Phylogenomic Analysis of Lactobacillus curvatus Reveals Two Lineages Distinguished by Genes for Fermenting Plant-Derived Carbohydrates. Genome biology and evolution 10, 1516–1525 (2018).
- Chaillou, S. et al. Origin and ecological selection of core and food-specific bacterial communities associated with meat and seafood spoilage. The ISME journal 9, 1105–1118 (2015).
Tous les articles de l’équipe sont disponibles dans la collection FME-MICALIS sur HAL.
- Horizon Europe RIA DOMINO (coordination). Harnessing the microbial potential of fermented food for healthy & sustainable food systems. 2023-2028.
- ANR-21-CE21-0003-01 METASIMFOOD (coordination). Improving the quality, safety and sustainability of fermented vegetable food and fruit beverage using a knowledge-driven synthetic ecology and modelling approach. 2022-2025.
- ANR-16-CE21-0006-03 REDLOSSES (partner). REDucing food LOSSES by microbial spoilage prediction. 2016-2020.
- ANR-14-CE20-0004-01 BLACHP (Partner). LACtic Bacteria combined with High Pressure for a sustainable stabilization process of refrigerated meat products. 2015-2018.
- ANR-ALIA ECOBIOPRO (coordination). Exploration of microbial ecosystems of fish and meat products. Effects of bioprotective cultures. 2010-2014.
- FERMENT DU FUTUR AAP2023 SYNTHPLEX 2023-2025 (coordination).
- FERMENT DU FUTUR AAP2023 SYNERGY 2023-2025 (coordination).
- HOLOFLUX metaprogramme EGG-TO-MEAT 2020-2021 (partner)
- DIGIT-BIO metaprogramme FERMENTWIN 2024-2025 (partner)
- MEM metaprogramme VIROME ACCESS 2018-2020 (coordination)
- MEM metaprogramme COMETES 2010-2012 (coordination)
- EMOVOL (private) 2021-2025 (coordination).